Python вставляет массив numpy в базу данных sqlite3
Я пытаюсь сохранить массив numpy около 1000 поплавков в базе данных sqlite3, но я продолжаю получать ошибку "InterfaceError: параметр привязки к ошибкам 1 - возможно, неподдерживаемый тип".
У меня было впечатление, что тип данных BLOB может быть чем угодно, но он определенно не работает с массивом numpy. Вот что я пробовал:
import sqlite3 as sql
import numpy as np
con = sql.connect('test.bd',isolation_level=None)
cur = con.cursor()
cur.execute("CREATE TABLE foobar (id INTEGER PRIMARY KEY, array BLOB)")
cur.execute("INSERT INTO foobar VALUES (?,?)", (None,np.arange(0,500,0.5)))
con.commit()
Есть ли другой модуль, который я могу использовать для получения массива numpy в таблице? Или я могу преобразовать массив numpy в другую форму в Python (например, список или строку, которую я могу разделить), которую примет sqlite? Производительность не является приоритетом. Я просто хочу, чтобы он работает!
Спасибо!
Ответы
Ответ 1
Вы можете зарегистрировать новый тип данных array с помощью sqlite3:
import sqlite3
import numpy as np
import io
def adapt_array(arr):
"""
http://stackoverflow.com/a/31312102/190597 (SoulNibbler)
"""
out = io.BytesIO()
np.save(out, arr)
out.seek(0)
return sqlite3.Binary(out.read())
def convert_array(text):
out = io.BytesIO(text)
out.seek(0)
return np.load(out)
# Converts np.array to TEXT when inserting
sqlite3.register_adapter(np.ndarray, adapt_array)
# Converts TEXT to np.array when selecting
sqlite3.register_converter("array", convert_array)
x = np.arange(12).reshape(2,6)
con = sqlite3.connect(":memory:", detect_types=sqlite3.PARSE_DECLTYPES)
cur = con.cursor()
cur.execute("create table test (arr array)")
С помощью этой настройки вы можете просто вставить массив NumPy без изменения синтаксиса:
cur.execute("insert into test (arr) values (?)", (x, ))
И получить массив непосредственно из sqlite в виде массива NumPy:
cur.execute("select arr from test")
data = cur.fetchone()[0]
print(data)
# [[ 0 1 2 3 4 5]
# [ 6 7 8 9 10 11]]
print(type(data))
# <type 'numpy.ndarray'>
Ответ 2
Это работает для меня:
import sqlite3 as sql
import numpy as np
import json
con = sql.connect('test.db',isolation_level=None)
cur = con.cursor()
cur.execute("DROP TABLE FOOBAR")
cur.execute("CREATE TABLE foobar (id INTEGER PRIMARY KEY, array BLOB)")
cur.execute("INSERT INTO foobar VALUES (?,?)", (None, json.dumps(np.arange(0,500,0.5).tolist())))
con.commit()
cur.execute("SELECT * FROM FOOBAR")
data = cur.fetchall()
print data
data = cur.fetchall()
my_list = json.loads(data[0][1])
Ответ 3
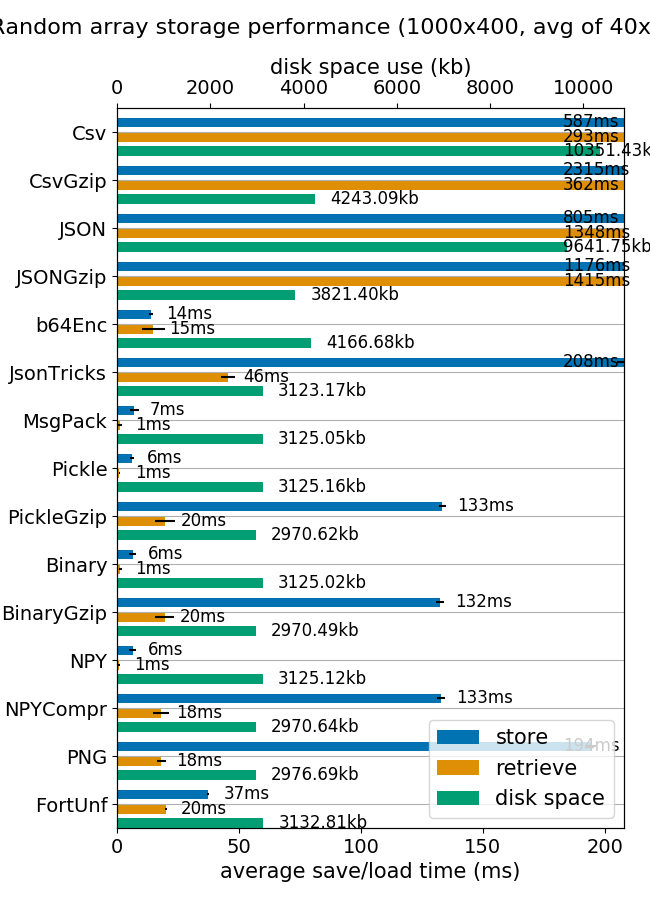
Я думаю, что формат matlab - это действительно удобный способ хранения и извлечения массивов. Это действительно быстро и дисков и памяти практически одинаковы.
![Load / Save / Disk Comparison]()
(изображение из тестов mverleg)
Но если по какой-либо причине вам нужно сохранить массивы в SQLite, я предлагаю добавить некоторые возможности сжатия.
Дополнительные строки из кода unutbu довольно просты
compressor = 'zlib' # zlib, bz2
def adapt_array(arr):
"""
http://stackoverflow.com/a/31312102/190597 (SoulNibbler)
"""
# zlib uses similar disk size that Matlab v5 .mat files
# bz2 compress 4 times zlib, but storing process is 20 times slower.
out = io.BytesIO()
np.save(out, arr)
out.seek(0)
return sqlite3.Binary(out.read().encode(compressor)) # zlib, bz2
def convert_array(text):
out = io.BytesIO(text)
out.seek(0)
out = io.BytesIO(out.read().decode(compressor))
return np.load(out)
Результаты тестирования с использованием базы данных MNIST:
$ ./test_MNIST.py
[69900]: 99% remain: 0 secs
Storing 70000 images in 379.9 secs
Retrieve 6990 images in 9.5 secs
$ ls -lh example.db
-rw-r--r-- 1 agp agp 69M sep 22 07:27 example.db
$ ls -lh mnist-original.mat
-rw-r--r-- 1 agp agp 53M sep 20 17:59 mnist-original.mat
'''
используя zlib, и
$ ./test_MNIST.py
[69900]: 99% remain: 12 secs
Storing 70000 images in 8536.2 secs
Retrieve 6990 images in 37.4 secs
$ ls -lh example.db
-rw-r--r-- 1 agp agp 19M sep 22 03:33 example.db
$ ls -lh mnist-original.mat
-rw-r--r-- 1 agp agp 53M sep 20 17:59 mnist-original.mat
используя bz2
Сравнивая формат Matlab V5 с bz2 на SQLite, сжатие bz2 составляет около 2.8, но время доступа довольно велико по сравнению с форматом Matlab (почти мгновенно против более 30 секунд). Может быть, подходит только для действительно огромных баз данных, где учебный процесс занимает больше времени, чем время доступа, или когда требуется, чтобы объем базы данных был как можно меньшим.
В заключение отметим, что отношение bipz/zlib составляет около 3,7, а zlib/matlab требует на 30% больше места.
Полный код, если вы хотите играть самостоятельно:
import sqlite3
import numpy as np
import io
compressor = 'zlib' # zlib, bz2
def adapt_array(arr):
"""
http://stackoverflow.com/a/31312102/190597 (SoulNibbler)
"""
# zlib uses similar disk size that Matlab v5 .mat files
# bz2 compress 4 times zlib, but storing process is 20 times slower.
out = io.BytesIO()
np.save(out, arr)
out.seek(0)
return sqlite3.Binary(out.read().encode(compressor)) # zlib, bz2
def convert_array(text):
out = io.BytesIO(text)
out.seek(0)
out = io.BytesIO(out.read().decode(compressor))
return np.load(out)
sqlite3.register_adapter(np.ndarray, adapt_array)
sqlite3.register_converter("array", convert_array)
dbname = 'example.db'
def test_save_sqlite_arrays():
"Load MNIST database (70000 samples) and store in a compressed SQLite db"
os.path.exists(dbname) and os.unlink(dbname)
con = sqlite3.connect(dbname, detect_types=sqlite3.PARSE_DECLTYPES)
cur = con.cursor()
cur.execute("create table test (idx integer primary key, X array, y integer );")
mnist = fetch_mldata('MNIST original')
X, y = mnist.data, mnist.target
m = X.shape[0]
t0 = time.time()
for i, x in enumerate(X):
cur.execute("insert into test (idx, X, y) values (?,?,?)",
(i, y, int(y[i])))
if not i % 100 and i > 0:
elapsed = time.time() - t0
remain = float(m - i) / i * elapsed
print "\r[%5d]: %3d%% remain: %d secs" % (i, 100 * i / m, remain),
sys.stdout.flush()
con.commit()
con.close()
elapsed = time.time() - t0
print
print "Storing %d images in %0.1f secs" % (m, elapsed)
def test_load_sqlite_arrays():
"Query MNIST SQLite database and load some samples"
con = sqlite3.connect(dbname, detect_types=sqlite3.PARSE_DECLTYPES)
cur = con.cursor()
# select all images labeled as '2'
t0 = time.time()
cur.execute('select idx, X, y from test where y = 2')
data = cur.fetchall()
elapsed = time.time() - t0
print "Retrieve %d images in %0.1f secs" % (len(data), elapsed)
if __name__ == '__main__':
test_save_sqlite_arrays()
test_load_sqlite_arrays()
Ответ 4
У Happy Leap Second это близко, но я продолжал получать автоматическое приведение к струне. Также, если вы прочитаете этот другой пост: веселые дебаты об использовании буфера или двоичного кода для передачи нетекстовых данных в sqlite, вы увидите, что документированный подход заключается в том, чтобы полностью избежать буфера и использовать этот кусок кода.
def adapt_array(arr):
out = io.BytesIO()
np.save(out, arr)
out.seek(0)
return sqlite3.Binary(out.read())
Я не очень тщательно тестировал это в Python 3, но, похоже, работает в Python 2.7
Ответ 5
Другие указанные методы не работают для меня. И, похоже, сейчас есть метод numpy.tobytes и numpy.fromstring (который работает с байтовыми строками), но не рекомендуется, и рекомендуемый метод - numpy.frombuffer.
import sqlite3
import numpy as np
sqlite3.register_adapter(np.array, adapt_array)
sqlite3.register_converter("array", convert_array)
Подойдя к мясу и картошке,
def adapt_array(arr):
return arr.tobytes()
def convert_array(text):
return np.frombuffer(text)
Я протестировал его в своем приложении, и он хорошо работает для меня на Python 3.7.3 и numpy 1.16.2
numpy.fromstring выдает те же выходные данные, что и DeprecationWarning: The binary mode of fromstring is deprecated, as it behaves surprisingly on unicode inputs. Use frombuffer instead DeprecationWarning: The binary mode of fromstring is deprecated, as it behaves surprisingly on unicode inputs. Use frombuffer instead